Eukaryotic stress-induced mutagenesis is limited by a local control of translesion synthesis.
Masłowska KH, Villafañez F, Laureti L, Iwai S, Pagès V


Le Centre de Recherche en Cancérologie de Marseille fête ses 50 ans ! -
Malgré des systèmes de réparation efficaces, il est fréquent que la fourche de réplication rencontre des lésions résiduelles. Les cellules disposent de deux stratégies de tolérance des lésions :
L’équilibre entre ces deux voies est très important puisqu’il définit le niveau de mutagenèse lors de la réplication d’un génome endommagé.
Nous avons développé une nouvelle technique qui nous permet d’insérer une lésion unique en un site spécifique du chromosome bactérien (Escherichia coli). Cette approche nous permet de suivre in vivo et de manière quantitative, la répartition entre synthèse translésionnelle (TLS) et contournement des dommages (DA) dans différents fonds génétiques, afin de déterminer les gènes qui régulent l’équilibre entre ces deux voies. Nous analysons également les intermédiaires de réplication précoces qui se forment au niveau de la lésion, par des approches moléculaires (qPCR, gel d’agarose en 2D, microscopie électronique), afin de définir les mécanismes impliqués dans les voies de tolérance des lésions.
Nous avons développé une approche similaire dans les cellules eucaryotes de levure (Saccharomyces cerevisiae). L’insertion d’une lésion unique dans le génome de cet organisme nous permet d’explorer les mécanismes de tolérance des dommages de l’ADN dans cet organisme.
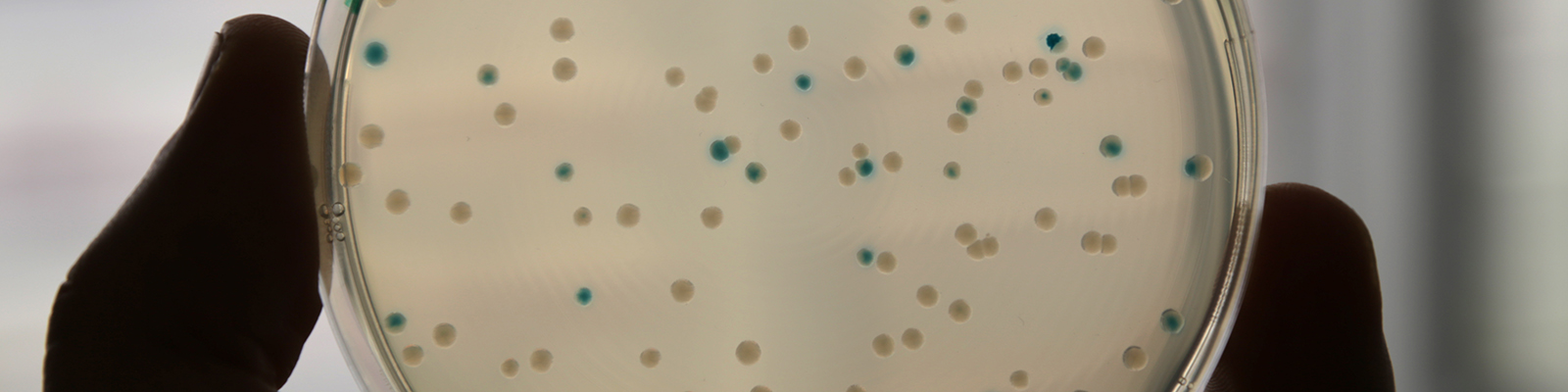 Colonies bactériennes : suite à l’insertion d’une lésion dans le génome, les colonies ayant répliqué la lésion (TLS) apparaissent bleues.
Colonies bactériennes : suite à l’insertion d’une lésion dans le génome, les colonies ayant répliqué la lésion (TLS) apparaissent bleues.

Les publications de l’équipe
Masłowska KH, Villafañez F, Laureti L, Iwai S, Pagès V
Laureti L, Lee L, Philippin G, Kahi M, Pagès V
Masłowska KH, Laureti L, Pagès V
Guervilly JH, Blin M, Laureti L, Baudelet E, Audebert S, Gaillard PH
Laureti L, Pagès V
Chrabaszcz É, Laureti L, Pagès V
Pagès V, Fuchs RP
Laureti L, Lee L, Philippin G, Pagès V
Naiman K, Pagès V, Fuchs RP
Pagès V
Laureti L, Demol J, Fuchs RP, Pagès V
Naiman K, Philippin G, Fuchs RP, Pagès V
Pagès V, Mazón G, Naiman K, Philippin G, Fuchs RP